این مقاله انگلیسی ISI در نشریه Ashpublications در 10 صفحه در سال 2013 منتشر شده و ترجمه آن 23 صفحه میباشد. کیفیت ترجمه این مقاله ویژه – طلایی ⭐️⭐️⭐️ بوده و به صورت کامل ترجمه شده است.
| دانلود رایگان مقاله انگلیسی + خرید ترجمه فارسی | |
| عنوان فارسی مقاله: |
تجزیه و تحلیل توالی ژنتیکی بیماری های خونریزی دهنده ارثی |
| عنوان انگلیسی مقاله: |
Genetic sequence analysis of inherited bleeding diseases |
|
|
|
| مشخصات مقاله انگلیسی | |
| فرمت مقاله انگلیسی | pdf و ورد تایپ شده با قابلیت ویرایش |
| سال انتشار | 2013 |
| تعداد صفحات مقاله انگلیسی | 10 صفحه با فرمت pdf |
| نوع مقاله | ISI |
| نوع نگارش | مقاله مروری (Review Article) |
| نوع ارائه مقاله | ژورنال |
| رشته های مرتبط با این مقاله | پزشکی و زیست شناسی |
| گرایش های مرتبط با این مقاله | ژنتیک، ژنتیک پزشکی، هماتولوژی |
| چاپ شده در مجله (ژورنال) | خون – BLOOD |
| ارائه شده از دانشگاه | گروه پاتوفیزیولوژی و پیوند، دانشگاه میلان، بنیاد لوئیجی ویلا، میلان، ایتالیا |
| نویسندگان | Flora Peyvandi – Tom Kunicki – David Lillicrap |
| شناسه شاپا یا ISSN | 0006-4971 |
| شناسه دیجیتال – doi | https://doi.org/10.1182/blood-2013-05-505511 |
| ایمپکت فاکتور(IF) مجله | 10.565 در سال 2019 |
| شاخص H_index مجله | 448 در سال 2020 |
| شاخص SJR مجله | 5.416 در سال 2019 |
| شاخص Q یا Quartile (چارک) | Q1 در سال 2019 |
| بیس | نیست ☓ |
| مدل مفهومی | ندارد ☓ |
| پرسشنامه | ندارد ☓ |
| متغیر | ندارد ☓ |
| رفرنس | دارای رفرنس در داخل متن و انتهای مقاله ✓ |
| کد محصول | 10979 |
| نشریه | Ashpublications |
| مشخصات و وضعیت ترجمه فارسی این مقاله | |
| فرمت ترجمه مقاله | pdf و ورد تایپ شده با قابلیت ویرایش |
| وضعیت ترجمه | انجام شده و آماده دانلود |
| کیفیت ترجمه | ویژه – طلایی ⭐️⭐️⭐️ |
| تعداد صفحات ترجمه تایپ شده با فرمت ورد با قابلیت ویرایش | 23 (2 صفحه رفرنس انگلیسی) صفحه با فونت 14 B Nazanin |
| ترجمه عناوین تصاویر و جداول | ترجمه شده است ✓ |
| ترجمه متون داخل تصاویر | ترجمه شده است ✓ |
| ترجمه متون داخل جداول | ترجمه شده است ✓ |
| ترجمه ضمیمه | ندارد ☓ |
| ترجمه پاورقی | ندارد ☓ |
| درج تصاویر در فایل ترجمه | درج شده است ✓ |
| درج جداول در فایل ترجمه | درج شده است ✓ |
| درج فرمولها و محاسبات در فایل ترجمه | ندارد ☓ |
| منابع داخل متن | به صورت عدد درج شده است ✓ |
| منابع انتهای متن | به صورت انگلیسی درج شده است ✓ |
| فهرست مطالب |
|
هموفیلی A و B هموفیلی B leiden ژنوتیپ های متفاوت – تست فاکتور VIII VWD نقایص نادر فاکتور کوآگولاسیون (انعقاد) طیف جهشی اختلالات خون ریزی دهنده ی نادر چالش های باقی مانده در ویژگی یابی ژنتیکی نقایص نادر فاکتور کوآگولاسیون (انعقادی) اختلالات خون ریزی دهنده ی ارثی مرتبط با پلاکت ها 1. واریانت های معمول با اثرات کوچک 2. واریانت های نادر با اثرات بزرگ تعیین خصوصیات اختلالات ارثی پلاکت اختلال پلاکت خانوادگی همراه با لوسمی میلوئیدی حاد سندرم هرمانسکی – پودلاک (HPS) ترومبوسیتوپنی 2، که تحت عنوان ترومبوسیتوپنی مرتبط با دمین تکرار آنکرین 26 (ANKRD26) نیز شناخته می شود سندرم ترومبوسیتوپنی همراه با فقدان رادیوس سندرم آلدریچ ویسکوت (WAS) استراتژی های ژنومی آینده برای تعیین خصوصیت اختلالات خون ریزی دهنده ی ارثی |
| بخشی از ترجمه |
|
ژن های کدکننده ی پروتئین های فاکتور انعقادی (کوآگولاسیون) در بین اولین ژن های انسانی قرار دارند که در طی 25 سال گذشته تعیین ویژگی شده اند. از آن زمان تا حالا، پیشرفت قابل توجهی در کاربردی سازی این اطلاعات برای 2 تا از معمول ترین اختلالات شدید خون ریزی دهنده ی ارثی، یعنی هموفیلی A و B صورت گرفته است. برای این اختلالات وابسته به X، تعیین خصوصیت ژنتیکی جهش های ایجاد کننده ی بیماری، حالا به درون استانداردهای مراقبتی اضافه شده است و اطلاعات ژنتیکی برای طبقه بندی ریسک عوارض درمانی مورد استفاده قرار گرفته اند. از روی داده پایگاه های الکترونیکی که بیشتر از 2100 جهش منحصر به فرد برای هموفیلی A و بیشتر از 1100 جهش برای هموفیلی B را به تفصیل بیان کرده اند، می توان نتیجه گرفت که این بیماری ها در بین گسترده ترین اختلالات ارثی در انسان ها قرار دارند که تعیین ویژگی شده اند. آزمایش ژنتیکی بیماری های خون ریزی دهنده ی نادر، همانطور که انتظارش می رود، پیشرفت کمی داشته است. به هرحال، اینجا بازهم، پایگاه داده های الکترونیکی جهش ایجاد شده اند و رهنمودهای عالی را برای کاربرد آنالیزهای ژنتیکی، به عنوان یک روش تاییدی برای تشخیص، ارائه کرده اند. اخیرا، پیشرفت هایی در زمینه ی شناسایی لوکوس های جهش یافته در یک طیفی از اختلالات ارثی پلاکت خون انجام شده است و این یافته ها آغاز به کاربرد برای تشخیص ژنتیکی این شرایط کرده اند. بررسی بیمارانی که فنوتیپ خون ریزی دهنده داشتند اما با استفاده از استراتژی های ژنوم گسترده تشخیص داده نشده بودند، ممکن است ژن های جدیدی را شناسایی کند که قبلا مشخص نشده بود که یک نقشی را در هموستازی دارند (خون. 2013، 122(20):3423-3431).
استراتژی های ژنومی آینده برای تعیین خصوصیت اختلالات خون ریزی دهنده ی ارثی همانطور که تکنولوژی های ژنومی به پیشرفت خود ادامه می دهند، پتانسیل این استراتژی ها برای الحاق به درون الگوریتم های تشخیصی نیز قطعا افزایش پیدا می کند. به هرحال، قبل از اینکه این امر بتواند انجام شود، یک تعدادی از ضمیمه های مهم به فراساختار تشخیصی اخیر ما باید اضافه شود. پایگاه داده هایی که به دقت برگزیده شده اند، مانند Reactome (www.reactome.org)، اطلاعات مهمی را فراهم می کنند که پزشکان و دانشمندان را قادر می سازد تا به اطلاعات درستی در مورد مسیرهای سلولی و تجسم روابط بین واریانت های توالی نادر و فنوتیپ های بالینی دست پیدا کنند. پایگاه داده ی GEN2PHEN (www.gen2phen.org) ازطریق یک همکاری بین مرکز ملی برای بیوانفورماتیک و موسسه ی بیوانفورماتیک اروپا ایجاد شده است و سایر تلاش ها منجر به ایجاد سایت لوکوس ژنوم مرجع (www.Irg-sequence.org) شده اند، این سیستم، یک توالی DNAیی ژنومی را ارائه می کند که نماینده ی یک ژن منفرد است که محتوای مرکزی آن هرگز تغییر نمی کند و یک لایه ی حاشیه ای قابل به روز رسانی دارد که شامل واریانت های توالی است. |
| بخشی از مقاله انگلیسی |
|
The genes encoding the coagulation factor proteins were among the first human genes to be characterized over 25 years ago. Since then, significant progress has been made in the translational application of this information for the 2 commonest severe inherited bleeding disorders, hemophilia A and B. For these X-linked disorders, genetic characterization of the disease-causing mutations is now incorporated into the standard of care and genetic information is used for risk stratification of treatment complications. With electronic databases detailing >2100 unique mutations for hemophilia A and >1100 mutations for hemophilia B, these diseases are among the most extensively characterized inherited diseases in humans. Experience with the genetics of the rare bleeding disorders is, as expected, less well advanced. However, here again, electronic mutation databases have been developed and provide excellent guidance for the application of genetic analysis as a confirmatory approach to diagnosis. Most recently, progress has also been made in identifying the mutant loci in a variety of inherited platelet disorders, and these findings are beginning to be applied to the genetic diagnosis of these conditions. Investigation of patients with bleeding phenotypes without a diagnosis, using genome-wide strategies, may identify novel genes not previously recognized as playing a role in hemostasis. (Blood. 2013; 122(20):3423-3431)
Future genomic strategies for the characterization of inherited bleeding disorders As genomic technologies continue to advance, the potential for incorporating these strategies into diagnostic algorithms will inevitably increase. However, before this can happen, a number of critical additions to our current diagnostic infrastructure must be developed. Carefully curated databases, such as Reactome (www.reactome. org), provide key information that will enable physicians and scientists to access relevant information on cellular pathways and to visualize the relationships between rare sequence variants and clinical phenotypes. The GEN2PHEN database (www.gen2phen.org), created through a collaboration between the National Center for Bioinformatics and European Bioinformatics Institute, and other efforts have resulted in the establishment of the Locus Reference Genome (www.lrg-sequence. org), a system that provides a genomic DNA sequence representing a single gene, whose core content never changes, with an updateable annotation layer that includes sequence variants. |
|
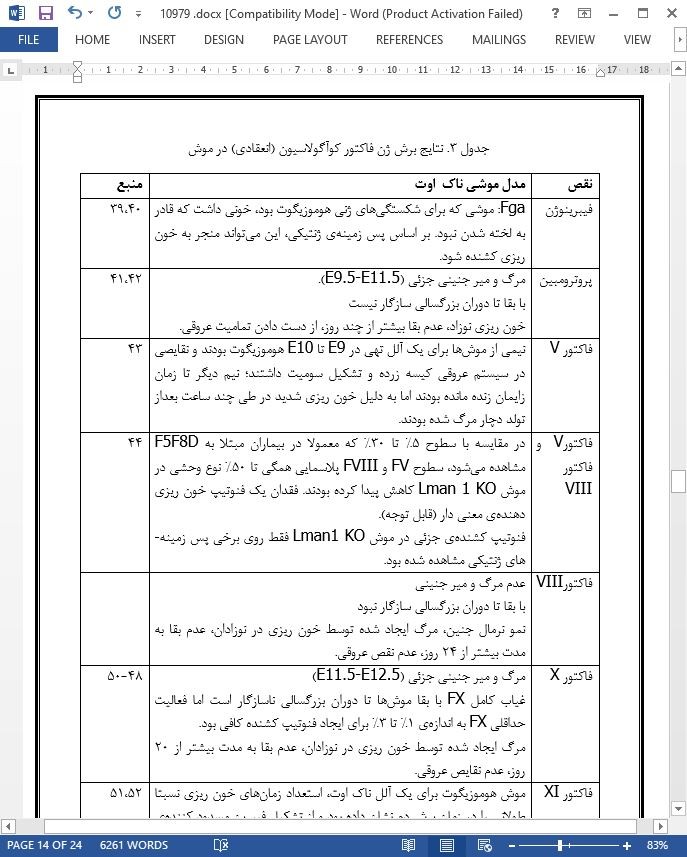
تصویری از مقاله ترجمه و تایپ شده در نرم افزار ورد |
|
|
| دانلود رایگان مقاله انگلیسی + خرید ترجمه فارسی | |
| عنوان فارسی مقاله: |
تجزیه و تحلیل توالی ژنتیکی بیماری های خونریزی دهنده ارثی |
| عنوان انگلیسی مقاله: |
Genetic sequence analysis of inherited bleeding diseases |
|
|
|