این مقاله انگلیسی ISI در نشریه Frontiersin در 11 صفحه در سال 2014 منتشر شده و ترجمه آن 22 صفحه میباشد. کیفیت ترجمه این مقاله ویژه – طلایی ⭐️⭐️⭐️ بوده و به صورت کامل ترجمه شده است.
| دانلود رایگان مقاله انگلیسی + خرید ترجمه فارسی | |
| عنوان فارسی مقاله: |
بازسازی جامع و تجسم شبکه های تنظیمی غیر کد کننده در انسان |
| عنوان انگلیسی مقاله: |
Comprehensive reconstruction and visualization of non-coding regulatory networks in human |
|
|
|
| مشخصات مقاله انگلیسی | |
| فرمت مقاله انگلیسی | pdf و ورد تایپ شده با قابلیت ویرایش |
| سال انتشار | 2014 |
| تعداد صفحات مقاله انگلیسی | 11 صفحه با فرمت pdf |
| نوع مقاله | ISI |
| نوع نگارش | Methods Article |
| نوع ارائه مقاله | ژورنال |
| رشته های مرتبط با این مقاله | زیست شناسی |
| گرایش های مرتبط با این مقاله | ژنتیک ، علوم سلولی و مولکولی ، میکروبیولوژی |
| چاپ شده در مجله (ژورنال) | مهندسی زیستی و بیوتکنولوژی – Bioengineering and Biotechnology |
| کلمات کلیدی | میکرو RNA، IncRNAs، RNA غیر کد کننده، شبکه ها، Cytospace، بیان ژن |
| کلمات کلیدی انگلیسی | microRNAs – lncRNAs – non-coding RNAs – networks – cytoscape – gene expression |
| ارائه شده از دانشگاه | گروه علوم کامپیوتر، دانشگاه ورونا، ایتالیا |
| نویسندگان | Vincenzo Bonnici – Francesco Russo – Nicola Bombieri – Alfredo Pulvirenti – Rosalba Giugno |
| شناسه دیجیتال – doi | https://doi.org/10.3389/fbioe.2014.00069 |
| بیس | نیست ☓ |
| مدل مفهومی | ندارد ☓ |
| پرسشنامه | ندارد ☓ |
| متغیر | ندارد ☓ |
| رفرنس | دارای رفرنس در داخل متن و انتهای مقاله ✓ |
| کد محصول | 11384 |
| لینک مقاله در سایت مرجع | لینک این مقاله در سایت Frontiersin |
| نشریه | Frontiersin |
| مشخصات و وضعیت ترجمه فارسی این مقاله | |
| فرمت ترجمه مقاله | pdf و ورد تایپ شده با قابلیت ویرایش |
| وضعیت ترجمه | انجام شده و آماده دانلود |
| کیفیت ترجمه | ویژه – طلایی ⭐️⭐️⭐️ |
| تعداد صفحات ترجمه تایپ شده با فرمت ورد با قابلیت ویرایش | 22 (2 صفحه رفرنس انگلیسی) صفحه با فونت 14 B Nazanin |
| ترجمه عناوین تصاویر و جداول | ترجمه شده است ✓ |
| ترجمه متون داخل تصاویر | ترجمه شده است ✓ |
| ترجمه متون داخل جداول | ترجمه شده است ✓ |
| ترجمه ضمیمه | ندارد ☓ |
| ترجمه پاورقی | ندارد ☓ |
| درج تصاویر در فایل ترجمه | درج شده است ✓ |
| درج جداول در فایل ترجمه | درج شده است ✓ |
| درج فرمولها و محاسبات در فایل ترجمه | ندارد ☓ |
| منابع داخل متن | ترجمه شده است ✓ |
| منابع انتهای متن | به صورت انگلیسی درج شده است ✓ |
| فهرست مطالب |
|
1-مقدمه 2-ساخت و محتوا 2-1 منبع داده 2-2 طرح داده 3-کاربرد 3-1 رابط Cytospace 3-2 رابط وب 3-3 رابط خط فرمان 4-نتیجه گیری |
| بخشی از ترجمه |
|
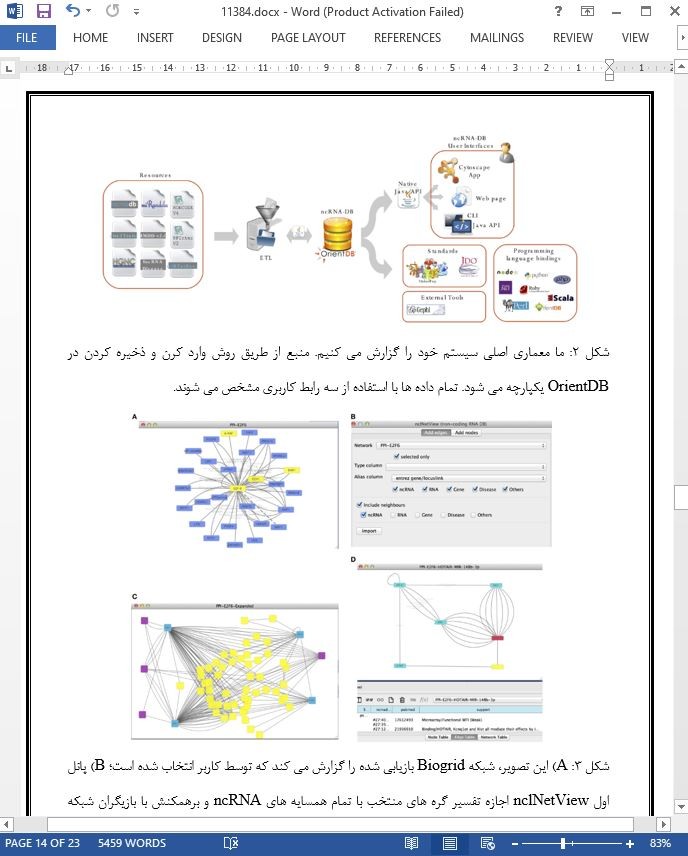
توجه تحقیقات معطوف به درک نقش عملیاتی RNAهای غیرکدکننده (ncRNAs) شده است. بسیاری از مطالعات، نقش عدم تنظیمی آنها در سرطان و دیگر اختلالات انسانی را نشان داده اند. ncRNAها در مایعات خارج سلولی بدن انسان وجود دارند، از جمله در سرم و پلاسما، و توانایی زیادی به عنوان نشانگر زیستی غیرتهاجمی دارند. اما، RNAهای غیرکدکننده اخیرا کشف شده اند و پایگاه داده جامعی که تمام آنها را دربرداشته باشد، وجود ندارد. بازسازی و تجسم شبکه برهمکنش های ncRNA، مراحل مهمی در درک مکانیزمهای تنظیمی آنها در سیستم های پیچیده هستند. این کار، یک پایگاه داده ncRNA-DB به نام NoSQL را ارائه می کند که تعامل داده های ncRNA را از مخازن آنلاین بزرگ با هم یکپارچه می کند. ncRNA-DB در http://ncnadb.scienze.it/ncrnadb ارائه شده است و مجهز به سه سطح است: مبتنی بر وب، خط فرمان و برنامه کاربردی Cytospace به نام nclNetView. با دستیابی به یک منبع، کاربران می توانند ncRNAها و برهمکنش های آنها را جستجو کنند و شبکه ای با تمام ncRNAها و بیماری های مرتبط بسازند و از تمام ویژگیهای بصری و کاوش موجود در Cytospace استفاده کنند.
1-مقدمه بعد از تعیین توالی ژنوم انسان، مشخص شد که فقط 20000 ژن کد کننده پروتئین هستند، در حالی که بیش از 98 درصد تمام ژن ها، RNAهای کد کننده غیرپروتئینی ترجمه نشده (ncRNAs) هستند (کنسرسیوم پروژه encode، 2012). معمولا، ncRNAها براساس طول خود به دو گروه تقسیم می شوند: cnRNAهای کوتاه که از کمتر از 200 نوکلئوتید تشکیل می شوند، و RNAهای غیر کد کننده طولانی (lncRNAs) که اندازه آنها از 200 نوکلئوتید تا 100kb متغیر است (ماتیک 2001).
4-نتیجه گیری در این مقاله، ما ncRNA-DB را ارائه کردیم که پایگاه داده یکپارچه ای است که دانش مربوط به ncRNAها، ژن ها و بیماری های مربوطه را ذخیره می کند. این سیستم در پایگاه داده NoSQL به نام OrientDB اجرا شده است و داده های ورودی از چندین منبع مهم را ذخیره می کند از جمله HGNC، lncRNAdb، cirx2Traita، HMDD، lncRNADiseases، miRandola، miRTarBase، NON-CODE و NPInter. ncRNA-DB را می توان از طریق سه رابط جستجو کرد. برنامه کاربردی Cytoscape، به نام nCINetView که اجازه تفسیر شبکه های زیستی با دانش ncRNA را می دهد. برنامه کاربردی وب و رابط خط فرمان که به کاربران اجازه جستجوی ncRNA-DB و استخراج اطلاعات در قالب متنی را می دهد. هدف از سیستم پیشنهادی، ارائه دسترسی جامع به تمام دانش موجود در ادبیات درباره ncRNA و بیماری مربوطه است. به عنوان یک ویژگی کلیدی، هدف داده های یکپارچه، کاهش دادن مساله نمادهای مختلف به کار رفته توسط منابع مختلف است. ncRNA-DB در http://ncrnadb.scienze.univr.it/ncrnadb قابل دسترسی است. |
| بخشی از مقاله انگلیسی |
|
Research attention has been powered to understand the functional roles of non-coding RNAs (ncRNAs). Many studies have demonstrated their deregulation in cancer and other human disorders. ncRNAs are also present in extracellular human body fluids such as serum and plasma, giving them a great potential as non-invasive biomarkers. However, non-coding RNAs have been relatively recently discovered and a comprehensive database including all of them is still missing. Reconstructing and visualizing the network of ncRNAs interactions are important steps to understand their regulatory mechanism in complex systems. This work presents ncRNA-DB, a NoSQL database that integrates ncRNAs data interactions from a large number of well established on-line repositories. The interactions involve RNA, DNA, proteins, and diseases. ncRNA-DB is available at http://ncrnadb.scienze.univr.it/ncrnadb/. It is equipped with three interfaces: web based, command-line, and a Cytoscape app called ncINetView. By accessing only one resource, users can search for ncRNAs and their interactions, build a network annotated with all known ncRNAs and associated diseases, and use all visual and mining features available in Cytoscape.
1. INTRODUCTION After the sequencing of the human genome, it became evident that only 20,000 genes are protein-coding, while over 98% of all genes are untranslated non-protein-coding RNAs (ncRNAs) (ENCODE Project Consortium, 2012). During the last years, thousands of ncRNAs have been identified in the eukaryotic transcriptome (Khalil et al., 2009; Bu et al., 2011). Usually, ncRNAs are divided into two groups according to their length: short ncRNAs, consisting of <200 nucleotides, and long non-coding RNAs (lncRNAs), whose size ranges from 200 nucleotides up to 100 kb (Mattick, 2001).
4. CONCLUSION In this paper, we have presented ncRNA-DB, an integrated database storing knowledge concerning ncRNAs, genes, and associated diseases. The system has been implemented within the NoSQL database OrientDB. It stores data coming from several leading resources such as HGNC, lncRNAdb, circ2Traits, HMDD, lncRNADiseases, miRandola, miRTarBase, NON-CODE, and NPInter. ncRNA-DB can be queried trough three interfaces. A Cytoscape App, named ncINetView, allows to annotate biological networks with ncRNA knowledge. A web app and a command-line interface, which allows users to query the ncRNA-DB and to extract information in a text format. The aim of the proposed system is to give a comprehensive access to all the knowledge available in the literature concerning ncRNAs and associated diseases. As a key characteristics, the integrated data aim to reduce the problem of different nomenclatures used by different sources. |
|
تصویری از مقاله ترجمه و تایپ شده در نرم افزار ورد |
|
|
| دانلود رایگان مقاله انگلیسی + خرید ترجمه فارسی | |
| عنوان فارسی مقاله: |
بازسازی جامع و تجسم شبکه های تنظیمی غیر کد کننده در انسان |
| عنوان انگلیسی مقاله: |
Comprehensive reconstruction and visualization of non-coding regulatory networks in human |
|
|
|