این مقاله انگلیسی ISI در نشریه NATURE در 11 صفحه در سال 2017 منتشر شده و ترجمه آن 26 صفحه میباشد. کیفیت ترجمه این مقاله ویژه – طلایی ⭐️⭐️⭐️ بوده و به صورت کامل ترجمه شده است.
| دانلود رایگان مقاله انگلیسی + خرید ترجمه فارسی | |
| عنوان فارسی مقاله: |
بهینه سازی ، تمایل و استفاده از کدون در ترجمه و تجزیه mRNA |
| عنوان انگلیسی مقاله: |
Codon optimality, bias and usage in translation and mRNA decay |
|
|
|
| مشخصات مقاله انگلیسی (PDF) | |
| سال انتشار مقاله | 2017 |
| تعداد صفحات مقاله انگلیسی | 11 صفحه با فرمت pdf |
| نوع مقاله | ISI |
| نوع نگارش | مقاله مروری (Review Article) |
| نوع ارائه مقاله | ژورنال |
| رشته های مرتبط با این مقاله | زیست شناسی |
| گرایش های مرتبط با این مقاله | علوم سلولی و مولکولی و ژنتیک |
| چاپ شده در مجله (ژورنال) | بررسی طبیعت بیولوژی سلول های مولکولی – Nature Reviews Molecular Cell Biology |
| ارائه شده از دانشگاه | ایالات متحده آمریکا |
| نمایه (index) | Scopus – Master journals – MedLine |
| نویسندگان | Gavin Hanson and Jeff Coller |
| شناسه شاپا یا ISSN | ISSN 1471-0080 |
| شناسه دیجیتال – doi | https://doi.org/10.1038/nrm.2017.91 |
| ایمپکت فاکتور(IF) مجله | 38.423 در سال 2018 |
| شاخص H_index مجله | 386 در سال 2018 |
| شاخص SJR مجله | 30.397 در سال 2018 |
| شاخص Q یا Quartile (چارک) | Q1 در سال 2018 |
| بیس | نیست ☓ |
| مدل مفهومی | ندارد ☓ |
| پرسشنامه | ندارد ☓ |
| متغیر | ندارد ☓ |
| رفرنس | دارد ✓ |
| کد محصول | 9682 |
| لینک مقاله در سایت مرجع | لینک این مقاله در سایت NATURE |
| نشریه NATURE | |
| مشخصات و وضعیت ترجمه فارسی این مقاله (Word) | |
| وضعیت ترجمه | انجام شده و آماده دانلود در فایل ورد و PDF |
| کیفیت ترجمه | ویژه – طلایی ⭐️⭐️⭐️ |
| تعداد صفحات ترجمه تایپ شده با فرمت ورد با قابلیت ویرایش | 26 صفحه با فونت 14 B Nazanin |
| ترجمه عناوین تصاویر | ترجمه شده است ✓ |
| ترجمه متون داخل تصاویر | ترجمه شده است ✓ |
| درج تصاویر در فایل ترجمه | درج شده است ✓ |
| منابع داخل متن | به صورت عدد درج شده است ✓ |
| فهرست مطالب |
|
چکیده بخش A ریبوزوم فریم خوانش باز ( ORF) شاخص تطبیق tRNA (tAI) ریبونوکلئوپروتئین (RNP) 80S پلی زوم کدون ها امتداد ترجمه را تحت تاثیر قرار می دهند کارایی ترجمه پروتئومیکس تفنگی توالی کدکننده ی DNA تاخوردگی پروتئین شاخص انطباق کدون ( CAI) استفاده از کدون، ثبات mRNA را کنترل می کند قطبیت اثرات کدون ارتباط عملکردی تغییرات در منابع tRNA به حفظ هموستازی سلولی کمک می کند استفاده از کدون و تکثیر سلولی نتیجه گیری و دیدگاه های آینده |
| بخشی از ترجمه |
|
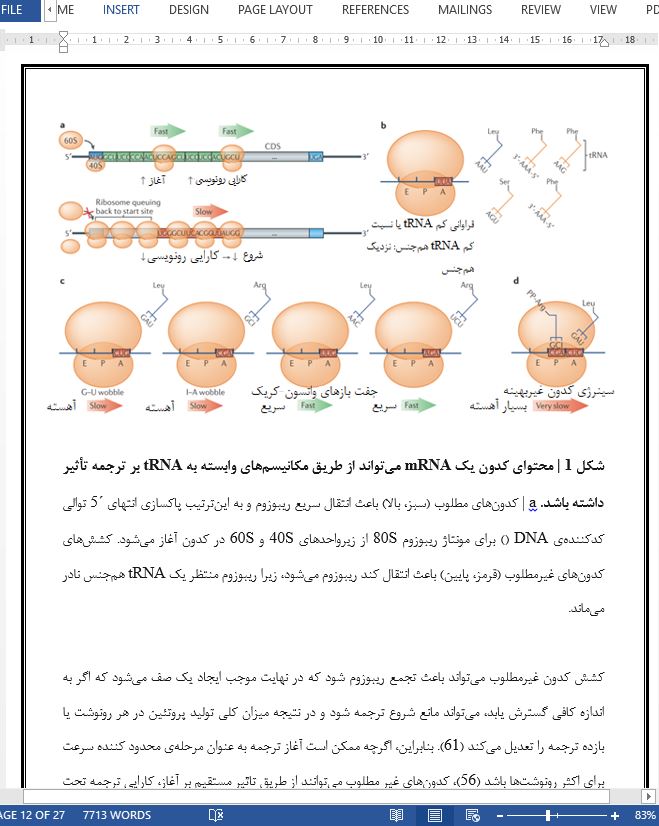
چکیده ظهور پروفایلینگ ریبوزوم و سایر ابزارها برای بررسی ترجمه mRNA نشان داده است که تمایل کدونی – استفاده نادرست از کدون های مترادف در ترنسکریپتوم – به عنوان یک کد ژنتیکی ثانویه عمل می کند: کدی که کارایی تولید پروتئین، صحت ترجمه و متابولیسم mRNA ها را هدایت می کند. پیشرفت های اخیر در درک ما از تخریب mRNA ، نشان دهنده ی یک اتصال تنگاتنگ بین پویایی ریبوزوم و ثبات رونوشت های mRNA است؛ این اتصال باعث گنجاندن تمایل کدونی در مفهوم بهینگی کدون یا اثراتی که کدون های خاص و غلظت های tRNA بر کارایی و درستی تشکیلات ترجمه می گذارند می شود. در این بررسی، ابتدا شواهد مربوط به اثرات وابسته به کدون بر ترجمه مورد بحث قرار می گیرد، که در ابتدا در مورد مکانیسم پایه ای بحث می شود که از طریق آن، اختلال ترجمه می تواند کارایی ترجمه، تاخوردگی پروتئین و ثبات رونوشت را تحت تأثیر قرار دهد. پس از آن، در مورد این بحث خواهیم کرد که چگونه اثرات کدون تحت تاثیر سلول قرار می گیرند تا باعث تقویت پروتئوم برای حفظ هموستازی، اجرای بیان ژن برنامه های رشد یا تمایز و بهینه سازی کارایی تولید پروتئین اتفاق بیافتد.
نتیجه گیری و دیدگاه های آینده در این مقاله مروری، در مورد پیامدهای متنوع عملکردی انحطاط در کد ژنتیکی بحث شده است. وجود کدون های متعدد برای یک اسید آمینه به سلول اجازه می دهد تا پیشرفت ریبوزوم را دقیقا تنظیم کند، زیرا با دستکاری غلظت های نسبی ایزوپذیرنده ی tRNA، یک پیام را ترجمه می کند. گونه های tRNA که در سلول کمیاب هستند، زمان بیشتری را برای رسیدن به جایگاه A ریبوزوم طی می کنند و لزوما باعث تاخیر در رونویسی ریبوزوم روی کدون هم جنس شده و در نتیجه بر صحت رونویسی و تاخوردگی پروتئین، مقدار پروتئین تولید شده و پایداری mRNA تاثیر می گذارد. اینکه دقیقا چگونه نسبت tRNA همجنس:نزدیک به هم جنس بر دینامیک طویل شدن و صحت رونویسی تاثیر می گذارد، به ویژه درک ویژگی های دقیق منبع tRNA تاثیرگذار بر برخی از فنوتیپ های وابسته به کدون که قبلا در مورد آن بحث کرده ایم، نیازمند مطالعه بیشتر است. اینکه محتوای کدونی تا چه حد می تواند بر هر کدام از این فرآیندها تاثیر بگذارد، به شدت به موقعیت کشش های بهینه سازی بالا و پایین کدون در رونوشت (16، 36) و محتوای توالی این کدون ها (66) و همچنین سایر عوامب شناسایی نشده بستگی دارد. |
| بخشی از مقاله انگلیسی |
|
Abstract The advent of ribosome profiling and other tools to probe mRNA translation has revealed that codon bias — the uneven use of synonymous codons in the transcriptome — serves as a secondary genetic code: a code that guides the efficiency of protein production, the fidelity of translation and the metabolism of mRNAs. Recent advancements in our understanding of mRNA decay have revealed a tight coupling between ribosome dynamics and the stability of mRNA transcripts; this coupling integrates codon bias into the concept of codon optimality, or the effects that specific codons and tRNA concentrations have on the efficiency and fidelity of the translation machinery. In this Review, we first discuss the evidence for codon-dependent effects on translation, beginning with the basic mechanisms through which translation perturbation can affect translation efficiency, protein folding and transcript stability. We then discuss how codon effects are leveraged by the cell to tailor the proteome to maintain homeostasis, execute specific gene expression programmes of growth or differentiation and optimize the efficiency of protein production.
Conclusions and future perspectives In this Review, we have discussed the diverse func‑ tional consequences of degeneracy in the genetic code. The existence of multiple codons for the same amino acid allows the cell to fine-tune the progress of the ribosome as it translates a message by manipulat‑ ing the relative concentrations of isoacceptor tRNAs. The tRNA species that are scarce in the cell will take longer to arrive at the A-site of the ribosome and neces‑ sarily cause delay in ribosome translocation over the cognate codon, thereby influencing the fidelity of translation and protein folding, the amount of pro‑ tein produced and the stability of the mRNA. Exactly how the cognate:near-cognate tRNA ratio influences elongation dynamics and the fidelity of the translation apparatus requires more research, especially to under‑ stand the more nuanced features of the tRNA pool that influence some of the codon-dependent phenotypes we have discussed. The extent to which codon content will influence any of these processes is crucially depend‑ ent on the location of high- or low-codon-optimality stretches within the transcript16,36 and the sequence context of these codons66, along with other factors that have not yet been identified. |
|
تصویری از مقاله ترجمه و تایپ شده در نرم افزار ورد |
|
|
| دانلود رایگان مقاله انگلیسی + خرید ترجمه فارسی | |
| عنوان فارسی مقاله: |
بهینه سازی ، تمایل و استفاده از کدون در ترجمه و تجزیه mRNA |
| عنوان انگلیسی مقاله: |
Codon optimality, bias and usage in translation and mRNA decay |
|
|
|