گروه آموزشی ترجمه فا اقدام به ارائه ترجمه مقاله با موضوع ” کاربرد ارائه مبتنی بر وب معنایی در شناسایی ژن عامل بیماری ” در قالب فایل ورد نموده است که شما عزیزان میتوانید پس از دانلود رایگان مقاله انگلیسی و نیز مطالعه نمونه ترجمه و سایر مشخصات، ترجمه را خریداری نمایید.
| دانلود رایگان مقاله انگلیسی + خرید ترجمه فارسی
|
|
| عنوان فارسی مقاله: |
شناسایی ژن های عامل بیماری با استفاده از ارائه مبتنی بر وب معنایی تلفیق دانش ژنومیکی و فنومیکی |
| عنوان انگلیسی مقاله: |
Identifying disease-causal genes using Semantic Web-based representation of integrated genomic and phenomic knowledge |
|
|
| مشخصات مقاله انگلیسی (PDF) | |
| سال انتشار | 2008 |
| تعداد صفحات مقاله انگلیسی | 13 صفحه با فرمت pdf |
| رشته های مرتبط با این مقاله | پزشکی و زیست شناسی |
| گرایش های مرتبط با این مقاله | بیوانفورماتیک، آسیب شناسی، ژنتیک پزشکی و ژنتیک |
| مجله | مجله انفورماتیک بیومدیکال – Journal of Biomedical Informatics |
| دانشگاه | گروه مهندسی پزشکی، دانشگاه سینسیناتی، ایالات متحده آمریکا |
| کلمات کلیدی | وب معنایی، رتبه بندی معنای هستی شناسی، یکپارچه سازی داده ها، بیوانفورماتیک، NLP |
| رفرنس | دارد |
| لینک مقاله در سایت مرجع | لینک این مقاله در نشریه Elsevier |
| نشریه | Elsevier |
| مشخصات و وضعیت ترجمه فارسی این مقاله (Word) | |
| تعداد صفحات ترجمه تایپ شده با فرمت ورد با قابلیت ویرایش و فونت 14 B Nazanin | 27 صفحه |
| ترجمه عناوین تصاویر و جداول | ترجمه نشده است |
| ترجمه متون داخل تصاویر | ترجمه نشده است |
| ترجمه متون داخل جداول | ترجمه نشده است |
| درج تصاویر در فایل ترجمه | درج شده است |
| درج جداول در فایل ترجمه | درج شده است |
| درج فرمولها و محاسبات در فایل ترجمه به صورت عکس | درج شده است |
- فهرست مطالب:
چکیده
1. مقدمه
2. مواد و روش ها
1.2 منابع دانش
2.1.1 منابع دانش ژنومیکی
2.1.2 منابع دانش فنومیکی
2.2 نقشه برداری ویژگی های بالینی برای پیدا کردن مفاهیم UMLS
2.3 نقشه برداری ویژگی های بالینی به ژن
2.4. تولید RDF
2.5 رتبه بندی در وب معنایی (SW)
2.5.1 محاسبه اهمیت منابع
2.5.2 اهمیت وزن ذهنیت (SW) و عینیت (OW)
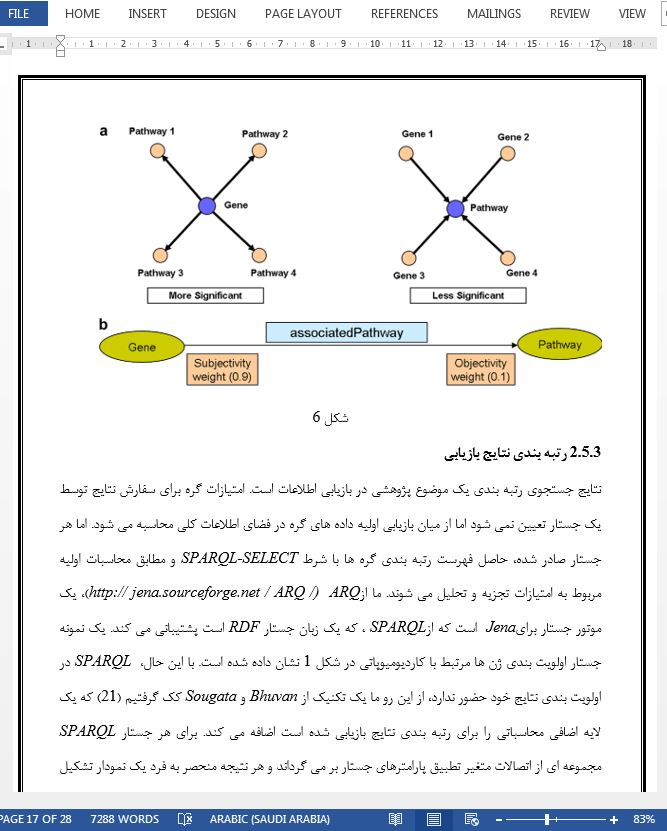
2.5.3 رتبه بندی نتایج بازیابی
3- نتایج
3.1. معیار روش
3.2. کاربرد
3.2.1. اولویت بندی ژن از CVD-مشمول مناطق ژنومی
4.مزایای استفاده از فن آوری های وب معنایی
4.1.ادغام انعطاف پذیر و جستار ژنومیکی وفنومیکی در شبکه
4.2.اضافه کردن متن از طریق وزن معنایی
4.3.توانایی بررسی منابع دیگر (به غیر از ژن ها) در RDF زیستی
5.بحث
6. نتیجه گیری
- بخشی از ترجمه:
6. نتیجه گیری
ما برای اولین بار در انسان اولویت بندی ژن بیماری را با ترکیبی از هر دو فنوتیپ موش و ویژگی های بالینی بیماری های انسانی از خلاصه بالینی OMIM انجام دادیم. علاوه بر این با چنین پوشش گسترده ای از منابع دانش استفاده شده، ما نیز برای اولین بار نشان دادیم که توانستیم از استانداردهای وب معنایی و تکنیک های اعمال شده با یک مشکل بیولوژیکی خاص به کار ببریم. استفاده مستقیم از RDF W3C و استانداردهای OWL برای ادغام دانش، برنامه های کاربردی شبکه های محوری را برای استخراج و بازیابی نتایج رتبه بندی شده با استفاده از زبان های نمودار جستار مثل SPARQLمورد تجزیه و تحلیل قرار دادیم. اگر چه در این مطالعه ما بر روی سیستم قلبی – عروقی تمرکز کردیم، رویکرد ما می تواند در هر گروه از ژن ها و یا بیماری به کار برده شود.
- بخشی از مقاله انگلیسی:
6. Conclusion
We have used for the first time in human disease gene prioritization a combination of both mouse phenotype and human disease clinical features from OMIM clinical synopsis. In addition to such extended coverage of knowledge sources used, we have also shown for the first time that one can leverage Semantic Web standards and techniques applied to a specific biological problem. The direct use of W3C’s RDF and OWL standards for knowledge integration, the application of network centrality analysis for mining and the retrieving of ranked results using graph query languages such as SPARQL. Although in this current study we focused on the cardiovascular system, our approach can be applied to any group of genes or diseases.
|
تصویری از مقاله ترجمه و تایپ شده در نرم افزار ورد |
|
|
| دانلود رایگان مقاله انگلیسی + خرید ترجمه فارسی
|
|
| عنوان فارسی مقاله: |
کاربرد ارائه مبتنی بر وب معنایی تلفیق دانش ژنومیکی و فنومیکی در شناسایی ژن عامل بیماری |
| عنوان انگلیسی مقاله: |
Identifying disease-causal genes using Semantic Web-based representation of integrated genomic and phenomic knowledge |
|
|